2.4 KiB
id, title, challengeType, forumTopicId, dashedName
| id | title | challengeType | forumTopicId | dashedName |
|---|---|---|---|---|
| 5900f49a1000cf542c50ffac | Problema 300: Enovelamento de proteínas | 5 | 301954 | problem-300-protein-folding |
--description--
De maneiras muito simplificada, podemos considerar as proteínas como strings consistindo em elementos hidrofóbicos (H) e polares (P), por exemplo, HHPPHHHPHHPH.
Para este problema, a orientação de uma proteína é importante; por exemplo, HPP é considerado diferente de PPH. Portanto, existem 2^n proteínas distintas compostas por n elementos.
Quando alguém encontra essas strings na natureza, elas estão sempre enoveladas de modo que o número de pontos de contato de H-H é tão grande quanto possível, pois isso é energeticamente vantajoso.
Como resultado, os elementos H tendem a se acumular na parte interna, com os elementos P ficando do lado de fora.
As proteínas naturais são enoveladas em três dimensões, é claro, mas só vamos considerar um enovelamento proteico em duas dimensões.
A figura abaixo mostra duas maneiras possíveis de o nosso exemplo de proteína poder ser enovelado (pontos de contato H-H são mostrados como pontos vermelhos).
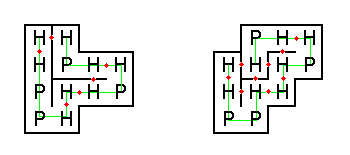
O enovelamento da esquerda tem apenas seis pontos de contato H-H e, por isso, nunca ocorreria naturalmente. Por outro lado, o enovelamento à direita tem nove pontos de contato H-H, o que é ideal para esta string.
Assumindo que os elementos H e P têm igual probabilidade de ocorrer em qualquer posição ao longo da string, o número médio de pontos de contato de H-H em um enovelamento ideal de uma string de proteína aleatória de comprimento 8 acaba sendo \frac{850}{2^8} = 3.3203125.
Qual é o número médio de pontos de contato de H-H em um enovelamento ideal de uma string de proteína aleatória de comprimento 15? Dê sua resposta usando tantas casas decimais quantas for necessário para um resultado exato.
--hints--
proteinFolding() deve retornar 8.0540771484375.
assert.strictEqual(proteinFolding(), 8.0540771484375);
--seed--
--seed-contents--
function proteinFolding() {
return true;
}
proteinFolding();
--solutions--
// solution required